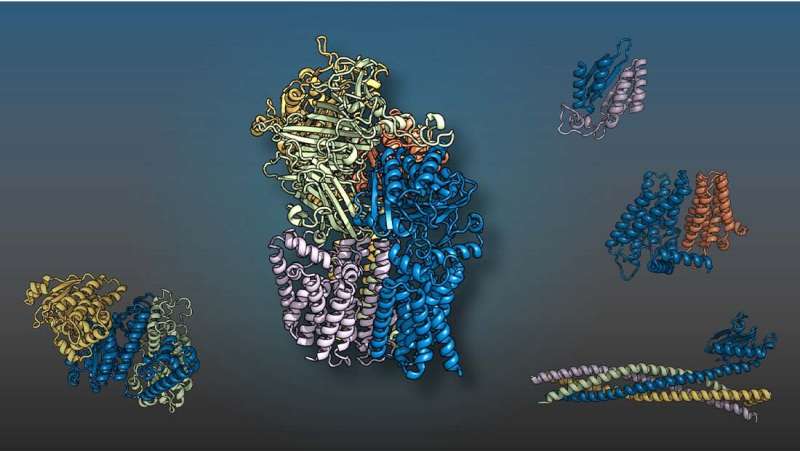
Las proteínas de levadura que se muestran en diferentes colores se combinan en complejos de dos, tres, cuatro y cinco miembros, como piezas de rompecabezas 3D, para realizar funciones celulares. Un equipo internacional dirigido por investigadores de UT Southwestern համալս University of Washington ha predicho las estructuras utilizando técnicas de inteligencia artificial. Préstamo: UT Southwestern Medical Center
Investigadores de la UT Southwestern համալս University of Washington dirigieron un equipo internacional que utilizó análisis evolutivo de inteligencia artificial (IA) para crear modelos 3D de interacciones de proteínas eucariotas. El estudio fue publicado Ciencias:, identificó por primera vez más de 100 complejos de proteínas potenciales, proporcionó modelos estructurales para más de 700 previamente inexplicables. La noción de compatibilidad de pares o grupos de proteínas para los procesos celulares puede conducir a muchos nuevos objetivos farmacológicos.
«Nuestros resultados representan un progreso significativo en la nueva era biología estructural en el que el cálculo juega un papel fundamental ”, dice Kean Kong, PhD, Asistente del Centro Eugene McDermott para el Crecimiento y el Desarrollo Humano, Nombramiento de Secundaria en Biofísica.
El Dr. Kong realizó la investigación con el Dr. David Baker պրոֆես, profesor de bioquímica, con el Dr. Kong, un mentor postdoctoral en la Universidad de Washington, antes de su contratación en UT Southwestern. El estudio tiene cuatro coautores, incluido el biólogo informático Jim Pay, Ph.D. de UT Southwest.
Las proteínas a menudo funcionan en pares o grupos, conocidos como complejos, para realizar todas las tareas necesarias para mantener vivo el cuerpo, explicó el Dr. Kong. Si bien algunas de estas interacciones están bien estudiadas, muchas siguen siendo un misterio. La creación de interacciones integrales o la descripción de un conjunto completo de interacciones moleculares en una célula arrojará luz sobre muchos aspectos fundamentales de la biología, lo que brindará a los investigadores un nuevo punto de partida para desarrollar fármacos que fomenten o desalienten esas interacciones. El Dr. Kong trabaja en el campo en evolución de la interactomía, que combina bioinformática y biología.
Hasta hace poco, el principal obstáculo para construir una interactomía era la incertidumbre sobre la estructura de muchas proteínas, que los científicos han estado tratando de resolver durante medio siglo. En 2020 և 2021, una empresa llamada DeepMind և El laboratorio del Dr. Baker lanzó de forma independiente dos tecnologías de inteligencia artificial, AlphaFold (AF) y RoseTTAFold (RF), que utilizan diferentes estrategias de pronóstico. proteína estructuras basadas en la secuencia de genes que las producen.
En un estudio en curso, el Dr. Kong y el Dr. Baker և y sus colegas desarrollaron estas herramientas para predecir la estructura de la inteligencia artificial mediante el modelado de múltiples complejos de proteínas de levadura. La levadura es un organismo modelo común para estudios biológicos básicos. Para encontrar las proteínas que tenían más probabilidades de interactuar, los investigadores primero observaron los genomas de hongos relacionados para encontrar genes que mutaron en huevos entrecruzados. Luego utilizaron dos tecnologías de inteligencia artificial para determinar si estas proteínas podrían encajar en estructuras 3D.
Su trabajo identificó 1505 posibles complejos de proteínas. 699 de ellos ya fueron probados estructuralmente, probando la utilidad de su método. Sin embargo, con solo datos experimentales limitados que respaldan 700 de las interacciones predichas, 106 nunca se describieron.
Para comprender mejor estos complejos desconocidos o mal caracterizados, los equipos de UT Southwestern en la Universidad de Washington trabajaron con socios de todo el mundo que ya han estudiado estas proteínas o proteínas similares. Al combinar modelos 3D creados por científicos en un estudio en curso con la información de los colaboradores, los equipos pudieron obtener nuevos conocimientos sobre los complejos de proteínas involucrados en el almacenamiento, procesamiento, estructura celular, sistemas de transporte, metabolismo y reparación del ADN de la información genética. otras areas. También exploraron los roles de proteínas cuyas funciones eran previamente desconocidas, basándose en interacciones recién descubiertas con otras proteínas bien caracterizadas.
«El trabajo descrito en nuestro nuevo estudio proporciona una base para estudios similares de interacción humana que, en última instancia, podrían ayudar a desarrollar nuevos tratamientos para las enfermedades humanas», añadió el Dr. Kong.
El Dr. Kong señaló que las estructuras proteicas complejas predichas resultantes de este estudio están disponibles para descargar ModeloArchivo:. Estas estructuras, y otras creadas con esta tecnología en investigaciones futuras, serán una rica fuente de preguntas de investigación en los próximos años, dijo.
El Dr. Kong es un científico de la Southwestern Medical Foundation en el campo de la investigación biomédica. Otros investigadores de UTSW que han contribuido a este estudio incluyen a Jing Zhang և Joseph Rizon, Ph.D., quien dirige el Departamento de Bioquímica en Virginia Lazenby O’Hara.
Ian R. Humphreys et al., Estructuras computadas de complejos proteicos eucariotas básicos Ciencias: (2021). DOI: 10.1126 / science.abm4805:
Está provisto
Centro médico de UT Southwest
Cita:Artificial Intelligence Predicts Protein Interactions (2021, 16 de noviembre), obtenido el 17 de noviembre de 2021: https://phys.org/news/2021-11-artificial-intelligence-successfully-protein-interactions.html
Este documento está protegido por derechos de autor. Ninguna parte de esta transacción puede reproducirse sin nuestro permiso por escrito, excepto para cualquier transacción o investigación privada. El contenido se proporciona solo con fines informativos.

Aficionado a los viajes. Lector exasperantemente humilde. Especialista en internet incurable
 Impulsse.la Complete News World
Impulsse.la Complete News World
